Illumina RNA Prep with Enrichment
Illumina RNA Prep with Enrichment
Быстрое, таргетное исследование большого числа таргетных генов с исключительной эффективностью захвата и равномерностью покрытия.
Illumina RNA Prep with Enrichment использует технологию On-Bead Tagmentation с последующей упрощенной единой гибридизацией для обеспечения исключительно быстрого рабочего процесса для подготовки библиотек на основе РНК и обогащений.
Быстрый и простой рабочий процесс для исследований любого масштаба
Подготовка и обогащение библиотек занимает менее 9 часов (пи этом работа руками составляет менее чем 2 часа). Протокол занимает в два раза меньше времени, чем TruSeq RNA Exome. Методы автоматизации, разработанные партнёрами и признанные компанией Illumina, станут доступны в ближайшее время.
Высококачественные данные из сложных образцов
Высокая чувствительность метода всего при 10 нг РНК из свежих или замороженных образцов или 20 нг РНК из деградированных образцов (таких как FFPE).
Доступность и сфокусированность
Выделение таргетных кодирующих областей транскриптома максимизирует возможности обнаружения на части глубины секвеенирования. Illumina RNA Prep with Enrichment разработан для обеспечения исключительной эффективности захвата и равномерности покрытия, минимизируя необходимую глубину секвенирования для точного обнаружения без отклонений. Для каждой реакции обогащения требуется только 2,5 мкл зондов.
Универсальность благодаря модульному строению панели
Возможность выбрать из множества предварительно созданных, проверенных пулов зондов или создать собственные зонды для максимальной универсальности. Мультиплексирование до 384 образцов за один запуск с новыми уникальными наборами двойных индексов C и D (скоро). Модульный дизайн поддерживает широкий спектр применений RNA-Seq в интересующих областях, позволяя проводить исследования по обнаружению аллель-специфических экспрессий, слияний, а также скрининг биомаркеров и многое другое.
Возможности работы с экзомом РНК
Быстрое таргетное исследование огромного числа таргетных генов, включая экзом РНК. Illumina RNA Prep with Enrichment можно использовать с панелью Illumina Exome Panel, которая имеет набор зондов с оптимальными характеристиками и обеспечивает полный охват кодирующих последовательностей РНК. Панель Illumina Exome включает более 425 000 зондов, каждый из которых построен от эталонного генома NCBI37/hg19, покрывая более 98% экзома RefSeq. Набор зондов захватывает более 214 000 таргетных участков, покрывая 21 415 генов, представляющих интерес.
Обнаружение респираторных вирусов
Respiratory Virus Oligo Panel обеспечивает точное обнаружение и определение распространенных респираторных вирусов, включая штаммы коронавирусов. Олигонуклеотидные зонды, используемые для таргетных обогащений посредством гибридного захватывания, остаются эффективными даже в сильно мутагенных областях, что позволяет исследовать быстро меняющиеся РНК-вирусы. Используйте Respiratory Virus Oligo Panel с Illumina RNA Prep with Enrichment.
Спецификации
|
Время анализа |
< 9 часов |
|---|---|
|
Время ручной работы |
< 2 часов |
| Тип нуклеиновой кислоты | РНК |
| Количество стартового материала |
10 нг РНК из свежих/замороженных образцов или 20 нг РНК из образцов FFPE |
| Особенности содержимого |
Захватывает кодирующий транскриптом при использовании с Illumina Exome Panel |
|
Описание |
Экономичное решение для обнаружения таргетных транскриптов из широкого спектра типов образцов, включая ткани FFPE и другие образцы низкого качества. |
| Мультиплексирование | До 384 уникальных двойных индексов (UDI) |
| Механизм действия |
Bead-linked transposome |
|
Для исследования каких видов подходит |
Человек, Вирус |
| Виды. Примечание | То, для каких видов подходит набор, зависит от используемой панели |
| Метод |
Секвенирование мРНК, таргетное обогащение, таргетное РНК-секвенирование |
| Класс вариантов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
| Совместимость | Набор совместим со всеми системами секвенирования Illumina, и тщательно проверен на системах NextSeq 500/550 и NovaSeq 6000 |
| Для работы с какими платформами подходит |
HiSeq 2500, HiSeq 3000, HiSeq 4000, iSeq 100, MiniSeq, MiSeq, NextSeq 2000, NextSeq 500, NextSeq 550, NovaSeq 6000 |
| Нестандартные типы образцов | кровь, ткани FFPE, низкое количество материалов образца |
| Стренд-специфичность набора | Не специфичен |
| Технология | Секвенирование |
| Возможность автоматизации | Автоматические станции пипетирования |
Рекомендации по проекту
|
Секвенатор |
Рекомендуемое количество образцов | Длина прочтения |
|---|---|---|
| NextSeq 550 |
Illumina RNA Prep with Enrichment + Exome Panel Mid Output: 5 High Output: 16 (из расчета 25 миллионов прочтений на образец) |
2 х 100 п.о. |
| NextSeq 2000 |
Illumina RNA Prep with Enrichment + Exome Panel P2: 16 P3: 40 (из расчета 25 миллионов прочтений на образец) |
2 х 100 п.о. |
| NovaSeq 6000 |
Illumina RNA Prep with Enrichment + Exome Panel SP: 32 S1: 64 S2: 164 S4: 384 (из расчета 25 миллионов прочтений на образец) |
2 х 100 п.о. |
| iSeq 100 |
Illumina RNA Prep with Enrichment + Respiratory Virus Panel 8 (из расчета 500 000 прочтений на образец) |
2 х 75 п.о. |
| MiSeq |
Illumina RNA Prep with Enrichment + Respiratory Virus Panel 1-50 (из расчета 500 000 прочтений на образец) |
2 х 75 п.о. |
| MiniSeq |
Illumina RNA Prep with Enrichment + Respiratory Virus Panel 8-50 (из расчета 500 000 прочтений на образец) |
2 х 75 п.о. |
| NextSeq 550 |
Illumina RNA Prep with Enrichment + Respiratory Virus Panel 260-384 (из расчета 500 000 прочтений на образец) |
2 х 75 п.о. |
Сравнение с другими наборами
|
|
Illumina Stranded Total RNA Prep with Ribo-Zero Plus |
TruSeq RNA Exome |
Illumina Stranded mRNA Prep |
|---|---|---|---|
|
Время анализа |
< 9 часов |
~2 дня | 6,5 часов |
|
Возможность автоматизации |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
Автоматические станции пипетирования |
|
Особенности содержимого |
Захватывает кодирующий транскриптом при использовании с Illumina Exome Panel |
Захватывает кодирующий транскриптом / РНК экзом |
Захватывает кодирующий транскриптом с информацией о цепях |
| Описание |
Экономичное решение для обнаружения таргетных транскриптов из широкого спектра типов образцов, включая ткани FFPE и другие образцы низкого качества. |
Предоставляет исследователям четкое и полное представление о кодирующем транскриптоме с точной информацией о цепях и менее жёсткими требованиями к стартовому материалу образца. |
Простое, экономически эффективное решение для анализа кодирующего транскриптома с точной информацией о цепях |
|
Время ручной работы |
< 2 часов |
~11 часов | < 3 часов |
| Количество стартового материала | 10 нг РНК из свежих/замороженных образцов или 20 нг РНК из образцов FFPE | 10 нг РНК из свежих/замороженных образцов или 20 нг РНК из образцов FFPE | 25-1000 нг РНК стандартного качества |
| Механизм действия | Bead-linked transposome | Биотинилированные зонды захвата, которые нацелены на кодирующую РНК. Не требует РНК с poly-А хвостами. | Захват PolyA, добавление адаптеров и индексов на основе лигирования |
| Метод |
Секвенирование мРНК, таргетное обогащение, таргетное РНК-секвенирование |
Секвенирование экзома, секвенирование мРНК |
Cеквенирование мРНК |
| Мультиплексирование | До 384 уникальных двойных индексов (UDI) |
До 24 одинарных, 96 комбинаторных (CD) двойных |
До 384 уникальных двойных индексов (UDI) |
| Класс вариантов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
Новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
Фьюжены генов, новые транскрипты, однонуклеотидные полиморфизмы (SNP), варианты транскриптов |
| Технология | Секвенирование | Секвенирование | Секвенирование |
Вспомогательные данные и показатели
Вирусы, обнаруженные с помощью Viral Enrichment Panel
| Human coronavirus 229E | Human parainfluenza virus 1 | Human bocavirus 4 NI strain HBoV4-NI-385 | Influenza A virus (A/Hong Kong/1073/99(H9N2)) |
| Human coronavirus NL63 | Human parainfluenza virus 2 | KI polyomavirus Stockholm 60 | Influenza A virus (A/Texas/50/2012(H3N2)) |
| Human coronavirus OC43 | Human parainfluenza virus 3 | WU Polyomavirus | Influenza A virus (A/Michigan/45/2015(H1N1)) |
| Human coronavirus HKU1 | Human parainfluenza virus 4a | Human parechovirus type 1 PicoBank/HPeV1/a | Influenza B virus (B/Lee/1940) |
| SARS-CoV-2 | Human metapneumovirus (CAN97-83) | Human parechovirus 6 | Influenza B virus (B/Wisconsin/01/2010) |
| Human adenovirus B1 | Respiratory syncytial virus (type A) | Human rhinovirus A89 | Influenza B virus (B/Brisbane/60/2008) |
| Human adenovirus C2 | Human Respiratory syncytial virus 9320 (type B) | Human rhinovirus C (strain 024) | Influenza B virus (B/Colorado/06/2017) |
| Human adenovirus E4 | Influenza A virus (A/Puerto Rico/8/1934(H1N1)) | Human rhinovirus B14 | Influenza B virus (B/Washington/02/2019) |
| Human bocavirus 1 (Primate bocaparvovirus 1 isolate st2) | Influenza A virus (A/Korea/426/1968(H2N2)) | Human enterovirus C104 strain: AK11 | Human control genes |
| Human bocavirus 2c PK isolate PK-5510 | Influenza A virus (A/New York/392/2004(H3N2)) | Human enterovirus C109 isolate NICA08-4327 | |
| Human bocavirus 3 | Influenza A virus (A/goose/Guangdong/1/1996(H5N1)) | Influenza A virus (A/Zhejiang/DTID-ZJU01/2013(H7N9)) |
Имеется 7800 зондов для выявления распространенных респираторных вирусов, недавних штаммов гриппа и SARS-CoV-2, а также зондов человека, которые выступают в качестве положительного контроля. Эти зонды представляют собой 80-нуклеотидные последовательности, которые располагаются очень близко друг к другу в процессе гибридизации, обеспечивая покрытие полного генома всех вирусов в панели.
Illumina RNA Prep with Enrichment обеспечивает быстрый рабочий процесс
Тагментация на шариках и одна 90-минутная гибридизация обеспечивают более быстрый рабочий процесс с меньшим количеством шагов по сравнению с TruSeq RNA Exome.
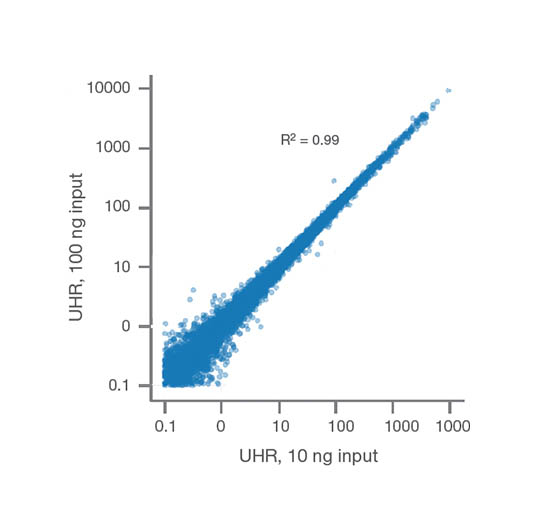
Данные высокого качества из малого количества стартового материала образцов
Illumina RNA Prep with Enrichment обеспечивает высокую согласованность данных у образцов с разным количеством стартового материала: 10 нг и 100 нг универсальной человеческой эталонной РНК (UHR). Библиотеки UHR-РНК секвенировали на системе NovaSeq 6000 с подвыборкой до 25 млн. кластеров на библиотеку. Данные были проанализированы с помощью приложения BaseSpace RNA-Seq Alignment v 1.1.1.
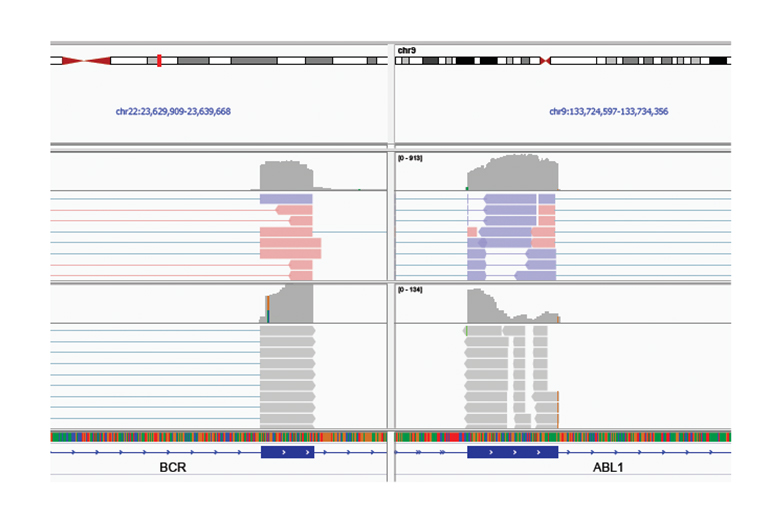
Обнаружение фьюжена гена BCR-ABL1
Библиотеки, приготовленные из 10 нг РНК из клеточной линии K-562 с использованием Illumina RNA Prepwith Enrichment и Illumina Exome Panel, привели к успешному обнаружению фьюжена гена BCR-ABL1. Верхний трек выравнивания показывает все риды, нижний трек показывает только риды, поддерживающие фьюжен гена BCR-ABL1.
Обнаружение фьюженов генов
| Фьюжен (источник) | RIN | Количество РНК | Обнаружение |
|---|---|---|---|
| BCR-ABL 1 (K-562) | 7,4 | 10 нг | 6/6 повторов (100%) |
| TPM3-NTRK1 (колоректальный рак) | 2,5 | 20 нг | 6/6 повторов (100%) |
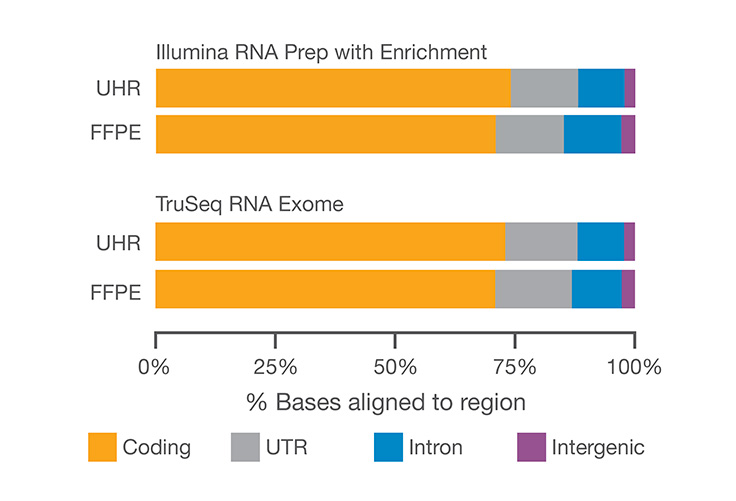
Покрытие кодирующих областей с помощью Illumina RNA Prep with Enrichment
Библиотеки, приготовленные из 10 нг универсальной человеческой эталонной РНК и 20 нг РНК из FFPE с использованием Illumina RNA Prep with Enrichment и Illumina Exome Panel, показывают более 85% данных,которые соответствуют кодирующим областям и нетранслируемым областям(UTR). Данные из библиотек TruSeq RNA Exome приведены для сравнения.